I vaccini a mRNA modificano il genoma umano. Ora lo dimostrano i primi studi
Trascrizione inversa intracellulare del vaccino Pfizer BioNTech COVID-19 mRNA BNT162b2 in vitro nella linea cellulare del fegato umano
1
Dipartimento di Scienze Cliniche, Università di Lund, 20502 Malmö, Svezia
2
Medicina delle infezioni, Dipartimento di Scienze Cliniche, Università di Lund, 22362 Lund, Svezia
*
Autore a cui indirizzare la corrispondenza.
Editore accademico: Stephen Malnick
Corr. Problemi Mol. Biol. 2022 , 44 (3), 1115-1126; https://doi.org/10.3390/cimb44030073 (registrazione DOI)
Ricevuto: 18 gennaio 2022
/
Revisionato: 19 febbraio 2022
/
Accettato: 23 febbraio 2022
/
Pubblicato: 25 febbraio 2022
(Questo articolo appartiene all'argomento Ricerca clinica, traslazionale e di base sulle malattie del fegato )
Studi
preclinici sul vaccino mRNA COVID-19 BNT162b2, sviluppato da Pfizer e
BioNTech, hanno mostrato effetti epatici reversibili negli animali che
hanno ricevuto l'iniezione di BNT162b2. Inoltre, uno studio recente ha
mostrato che l'RNA SARS-CoV-2 può essere trascritto inverso e integrato
nel genoma delle cellule umane. In questo studio, abbiamo studiato
l'effetto di BNT162b2 sulla linea cellulare di fegato umano Huh7 in
vitro. Le cellule Huh7 sono state esposte a BNT162b2 e la PCR
quantitativa è stata eseguita sull'RNA estratto dalle cellule. Abbiamo
rilevato livelli elevati di BNT162b2 nelle cellule Huh7 e cambiamenti
nell'espressione genica dell'elemento nucleare 1 lungo intervallato
(LINE-1), che è una trascrittasi inversa endogena. L'immunoistochimica
che utilizza il legame dell'anticorpo alla LINE-1 open reading frame-1
RNA-binding protein (ORFp1) su cellule Huh7 trattate con BNT162b2 ha
indicato una maggiore distribuzione del nucleo di LINE-1. La PCR sul
DNA genomico delle cellule Huh7 esposte a BNT162b2 ha amplificato la
sequenza di DNA unica di BNT162b2. I nostri risultati indicano un
rapido assorbimento di BNT162b2 nella linea cellulare del fegato umano
Huh7, portando a cambiamenti nell'espressione e nella distribuzione
della LINEA-1. Mostriamo anche che l'mRNA di BNT162b2 viene trascritto
intracellularmente nel DNA in appena 6 h dopo l'esposizione a BNT162b2.
Parole chiave:
Vaccino mRNA COVID-19 ; BNT162b2 ; fegato ; trascrizione inversa ; LINEA-1 ; Eh7
1. Introduzione
La
malattia da coronavirus 2019 (COVID-19) causata dalla sindrome
respiratoria acuta grave coronavirus 2 (SARS-CoV-2) è stata annunciata
dall'Organizzazione mondiale della sanità (OMS) come pandemia globale
l'11 marzo 2020 ed è emersa come una devastante crisi sanitaria . A
febbraio 2022, COVID-19 ha portato a oltre 430 milioni di casi di
infezione segnalati e 5,9 milioni di decessi in tutto il mondo [ 1 ]. Sono urgentemente necessari vaccini efficaci e sicuri per ridurre i tassi di morbilità e mortalità associati al COVID-19.
Sono
stati sviluppati diversi vaccini per COVID-19, con particolare
attenzione ai vaccini mRNA (di Pfizer-BioNTech e Moderna), ai vaccini
vettoriali adenovirali ricombinanti difettosi per la replicazione (di
Janssen-Johnson e Johnson, Astra-Zeneca, Sputnik-V e CanSino ) e vaccini
inattivati (di Sinopharm, Bharat Biotech e Sinovac). Il vaccino mRNA
presenta i vantaggi di essere flessibile ed efficiente nella
progettazione e produzione di immunogeni e attualmente numerosi vaccini
candidati sono in varie fasi di sviluppo e applicazione. In particolare,
il vaccino mRNA COVID-19 BNT162b2 sviluppato da Pfizer e BioNTech è
stato valutato in studi clinici di successo [ 2 , 3 , 4 ] e somministrato in campagne nazionali di vaccinazione COVID-19 in diverse regioni del mondo [ 5 , 6 , 7 , 8 ].
BNT162b2
è un vaccino a RNA (modRNA) incapsulato con nanoparticelle lipidiche
(LNP) e codifica per l'intera lunghezza della proteina spike (S)
SARS-CoV-2, modificata da due mutazioni della prolina per garantire una
conformazione pre-fusione antigenicamente ottimale , che imita il virus
intatto per suscitare anticorpi neutralizzanti il virus [ 3 ].
Coerentemente con gli studi clinici randomizzati, BNT162b2 ha mostrato
un'elevata efficienza in un'ampia gamma di esiti correlati a COVID-19 in
un contesto del mondo reale [ 5 ].
Tuttavia, permangono molte sfide, incluso il monitoraggio della
sicurezza e dell'efficacia a lungo termine del vaccino. Ciò garantisce
ulteriori valutazioni e indagini. Il profilo di sicurezza di BNT162b2 è
attualmente disponibile solo da studi clinici a breve termine. Sono
stati segnalati effetti avversi meno comuni di BNT162b2, tra cui
pericardite, aritmia, trombosi venosa profonda, embolia polmonare,
infarto del miocardio, emorragia intracranica e trombocitopenia [ 4 , 9 , 10 , 11 , 12 , 13 , 14 , 15 , 16 , 17 , 18 , 19 , 20 ]. Ci sono anche studi che riportano gli effetti avversi osservati in altri tipi di vaccini [ 21 , 22 , 23 , 24 ].
Per comprendere meglio i meccanismi alla base degli effetti avversi
correlati al vaccino, sono necessarie indagini cliniche e analisi
cellulari e molecolari.
Uno studio recente ha dimostrato che gli RNA SARS-CoV-2 possono essere trascritti e integrati nel genoma delle cellule umane [ 25 ].
Ciò fa sorgere la domanda se ciò possa verificarsi anche con BNT162b2,
che codifica per l'RNA SARS-CoV-2 parziale. Nei dati di farmacocinetica
forniti da Pfizer all'Agenzia europea per i medicinali (EMA), la
biodistribuzione di BNT162b2 è stata studiata nei topi e nei ratti
mediante iniezione intramuscolare con LNP radiomarcato e modRNA della
luciferasi. La radioattività è stata rilevata nella maggior parte dei
tessuti sin dal primo momento (0,25 h) ei risultati hanno mostrato che
il sito di iniezione e il fegato erano i principali siti di
distribuzione, con concentrazioni massime osservate a 8-48 h dopo la
dose [ 26 ].
Inoltre, negli animali che hanno ricevuto l'iniezione di BNT162b2, sono
stati osservati effetti epatici reversibili, tra cui ingrossamento del
fegato, vacuolazione, aumento dei livelli di gamma glutamil transferasi
(γGT) e aumento dei livelli di aspartato transaminasi (AST) e fosfatasi
alcalina (ALP) [ 26 ]. In precedenza sono stati riportati effetti epatici transitori indotti dai sistemi di rilascio di LNP [ 27 , 28 , 29 , 30 ], tuttavia, è stato anche dimostrato che l'LNP vuoto senza modRNA da solo non introduce alcun danno epatico significativo [ 27 ].
Pertanto, in questo studio, miriamo a esaminare l'effetto di BNT162b2
su una linea cellulare di fegato umano in vitro e studiare se BNT162b2
può essere trascritto inversamente nel DNA attraverso meccanismi
endogeni.
2. Materiali e metodi
2.1. Coltura cellulare
Le cellule Huh7 (JCRB Cell Bank, Osaka, Giappone) sono state coltivate a 37 ° C al 5% di CO 2 con terreno DMEM (HyClone, HYCLSH30243.01) integrato con siero bovino fetale al 10% ( v / v ) (Sigma-Aldrich, F7524 -500ML, Burlington, MA, USA) e 1% ( v / v )
di penicillina-streptomicina (HyClone, SV30010, Logan, UT, USA). Per il
trattamento con BNT162b2, le cellule Huh7 sono state seminate con una
densità di 200.000 cellule/pozzetto in piastre da 24 pozzetti. Il
vaccino mRNA BNT162b2 (Pfizer BioNTech, New York, NY, USA) è stato
diluito con iniezione sterile di cloruro di sodio allo 0,9%, USP in una
concentrazione finale di 100 μg/mL come descritto nelle linee guida del
produttore [ 31 ].
La sospensione di BNT162b2 è stata quindi aggiunta in terreni di
coltura cellulare per raggiungere concentrazioni finali di 0,5, 1,0 o
2,0 μg/mL. Le cellule Huh7 sono state incubate con o senza BNT162b2 per
6, 24 e 48 ore. Le cellule sono state lavate accuratamente con PBS e
raccolte mediante tripsinizzazione e conservate a -80 ° C fino a un
ulteriore utilizzo.
2.2. RT-QPCR IN TEMPO REALE
L'RNA
dalle cellule è stato estratto con RNeasy Plus Mini Kit (Qiagen, 74134,
Hilden, Germania) seguendo il protocollo del produttore. La RT-PCR è
stata eseguita utilizzando il kit RevertAid First Strand cDNA Synthesis
(Thermo Fisher Scientific, K1622, Waltham, MA, USA) seguendo il
protocollo del produttore. La qPCR in tempo reale è stata eseguita
utilizzando Maxima SYBR Green/ROX qPCR Master Mix (Thermo Fisher
Scientific, K0222, Waltham, MA, USA) con primer per BNT162b2, LINE-1 e geni di pulizia ACTB e GAPDH ( Tabella 1 ).
Tabella 1.
Sequenze di primer di RT-qPCR e PCR.
2.3. Immunofluorescenza e imaging confocale
Le
cellule Huh7 sono state coltivate in vetrini a otto camere (LAB-TEK,
154534, Santa Cruz, CA, USA) con una densità di 40.000 cellule/pozzetto,
con o senza BNT162b2 (0,5, 1 o 2 µg/mL) per 6 ore. L'immunoistochimica
è stata eseguita utilizzando l'anticorpo monoclonale di topo
anti-LINE-1 ORF1p primario (Merck, 3574308, Kenilworth, NJ, USA),
l'anticorpo secondario Cy3 Donkey anti-topo (Jackson ImmunoResearch,
West Grove, PA, USA) e Hoechst (Life tecnologie, 34850, Carlsbad, CA,
USA), seguendo il protocollo di Thermo Fisher (Waltham, MA, USA). Sono
state scattate due immagini per condizione utilizzando uno Zeiss LSM 800
e un obiettivo a immersione in olio 63X e l'intensità della colorazione
è stata quantificata sull'intera area cellulare individuale e sull'area
del nucleo su 15 cellule per immagine da ImageJ 1.53c. L'intensità
della colorazione LINE-1 per il citosol è stata calcolata sottraendo
l'intensità del nucleo da quella dell'intera cellula. A tutte le
immagini delle cellule è stato assegnato un numero casuale per evitare
pregiudizi. Per contrassegnare i nuclei (determinati dalla colorazione
di Hoechst) e le cellule intere (determinate dai bordi della
fluorescenza LINE-1), è stato utilizzato lo strumento di selezione a
mano libera. Queste aree sono state quindi misurate e l'intensità media
è stata utilizzata per confrontare i gruppi.
2.4. Purificazione del DNA genomico, amplificazione PCR, purificazione del gel di agarosio e sequenziamento di Sanger
Il
DNA genomico è stato estratto da pellet cellulari con tampone PBND (10
mM Tris-HCl pH 8,3, 50 mM KCl, 2,5 mM MgCl2, 0,45% NP-40, 0,45%
Tween-20) secondo il protocollo descritto in precedenza [ 32 ].
Per rimuovere l'RNA residuo dalla preparazione del DNA, RNasi (100 µ
g/mL, Qiagen, Hilden, Germania) è stata aggiunta alla preparazione del
DNA e incubata a 37 ° C per 3 ore, seguita da 5 minuti a 95 ° C. La PCR è
stata quindi eseguita utilizzando primer mirati a BNT162b2 (le sequenze
sono mostrate nella Tabella 1 ),
con il seguente programma: 5 minuti a 95 ° C, 35 cicli di 95 ° C per 30
s, 58 ° C per 30 s e 72 ° C per 1 minuto; infine, 72 °C per 5 min e 12
°C per 5 min. I prodotti della PCR sono stati eseguiti su p / v gel
di agarosio Le bande corrispondenti agli ampliconi della dimensione
prevista (444 bps) sono state tagliate e il DNA è stato estratto
utilizzando il kit di purificazione PCR QIAquick (Qiagen, 28104, Hilden,
Germania), seguendo le istruzioni del produttore. La sequenza
dell'amplicone del DNA è stata verificata mediante sequenziamento di
Sanger (Eurofins Genomics, Ebersberg, Germania).
Statistiche
di Student a due code t e l'ANOVA. I dati sono espressi come media ± SEM o ± SD. Le differenze con p < 0,05 sono considerate significative.
2.5. Dichiarazioni etiche
La linea cellulare Huh7 è stata ottenuta dalla banca cellulare giapponese Collection of Research Bioresources (JCRB).
3. Risultati
3.1. BNT162b2 entra nelle cellule Huh7 della linea cellulare del fegato umano ad alta efficienza
Per
determinare se BNT162b2 entra nelle cellule epatiche umane, abbiamo
esposto la linea cellulare epatica umana Huh7 a BNT162b2. In uno studio
precedente sulla cinetica di assorbimento dell'LNP nelle cellule Huh7,
la massima efficacia biologica dell'LNP è stata osservata tra 4 e 7 ore [
33 ].
Pertanto, nel nostro studio, le cellule Huh7 sono state coltivate con o
senza concentrazioni crescenti di BNT162b2 (0,5, 1,0 e 2,0 µ g/mL) per
6, 24 e 48 ore. L'RNA è stato estratto dalle cellule ed è stata eseguita
una reazione a catena della polimerasi a trascrizione inversa
quantitativa in tempo reale (RT-qPCR) utilizzando primer mirati alla
sequenza BNT162b2, come illustrato nella Figura 1 . La sequenza completa di BNT162b2 è disponibile pubblicamente [ 34 ]
e contiene un cappuccio a due nucleotidi; 5′- regione non tradotta
(UTR) che incorpora il 5′-UTR di un gene umano per l'α-globina; l'intera
lunghezza della proteina SARS-CoV-2 S con due mutazioni della prolina;
3′-UTR che incorpora il segmento mitocondriale umano 12S rRNA (mtRNR1) e
il segmento del gene AES/TLE5 umano con due mutazioni C→U; coda
poli(A). L'analisi dettagliata della sequenza della proteina S in
BNT162b2 ha rivelato 124 sequenze identiche al 100% alle sequenze
genomiche umane e tre sequenze con un solo disadattamento di nucleotide
(nt) in 19-26 nts ( Tabella S1, vedere Materiali supplementari ).
Per rilevare il livello di RNA di BNT162b2, abbiamo progettato primer
con primer forward situato nelle regioni della proteina SARS-CoV-2 S e
primer inverso in 3′-UTR, che consente il rilevamento dell'amplicone PCR
unico per BNT162b2 senza legame non specifico dei primer alle regioni
genomiche umane .
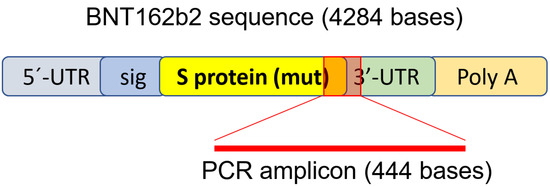
Figura 1.
Set di primer per PCR utilizzato per rilevare il livello di mRNA e la
trascrizione inversa di BNT162b2. L'illustrazione di BNT162b2 è stata
adattata dalla letteratura precedentemente descritta [ 34 ].
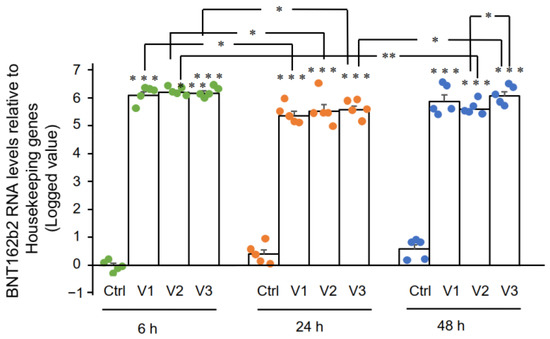
I
risultati di RT-qPCR hanno mostrato che le cellule Huh7 trattate con
BNT162b2 avevano livelli elevati di mRNA di BNT162b2 rispetto ai geni di
pulizia a 6, 24 e 48 ore ( Figura 2 , presentata in 2 -ΔΔCT loggati a
causa di livelli eccezionalmente alti). Le tre concentrazioni di
BNT162b2 hanno portato a livelli di mRNA di BNT162b2 intracellulari
simili in diversi momenti, tranne per il fatto che la differenza
significativa tra 1,0 e 2,0 µ g/mL è stata osservata a 48 ore. I livelli
di mRNA di BNT162b2 sono stati significativamente ridotti a 24 ore
rispetto a 6 ore, ma sono aumentati di nuovo a 48 ore.

Figura 2.
Livelli di mRNA di BNT162b2 nelle cellule Huh7 trattate con BNT162b2.
Le cellule Huh7 sono state trattate senza (Ctrl) o con 0,5 (V1), 1 (V2) e
2 µg/mL (V3) di BNT162b2 per 6 (punti verdi), 24 (punti arancioni) e 48
ore (punti blu) . L'RNA è stato purificato e la qPCR è stata eseguita
utilizzando primer mirati a BNT162b2. I livelli di RNA di BNT162b2 sono
presentati come valori registrati di 2 −ΔΔCT relativi ai geni di mantenimento GAPDH e ACTB . I risultati provengono da cinque esperimenti indipendenti ( n = 5). di Student a due code t . I dati sono espressi come media ± SEM. (* p < 0,05; ** p < 0,01; *** p < 0,001 rispetto al rispettivo controllo in ogni momento, o come indicato).
3.2. Effetto di BNT162b2 sulla trascrittasi inversa endogena umana elemento nucleare-1 lungo intervallato (LINE-1)
Qui abbiamo esaminato l'effetto di BNT162b2 sull'espressione LINE-1 .
La RT-qPCR è stata eseguita su RNA purificato da cellule Huh7 trattate
con BNT162b2 (0, 0,5, 1,0 e 2,0 µg/mL) per 6, 24 e 48 ore, utilizzando
primer mirati a LINE-1 . significativamente aumentata LINE-1 rispetto
al controllo è stata osservata a 6 ore di 2,0 µg/mL di BNT162b2, mentre
le concentrazioni di BNT162b2 inferiori hanno ridotto LINE-1 in tutti i punti temporali ( Figura 3 ).
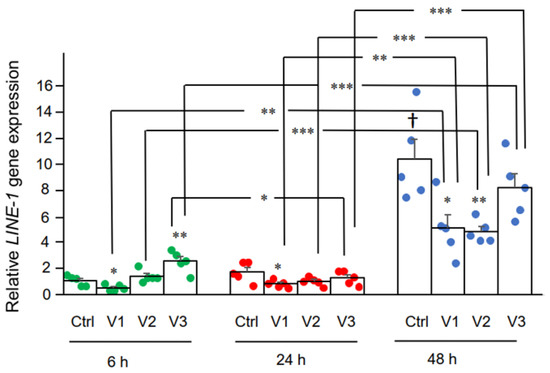
Figura 3.
LINE-1 nelle
cellule Huh7 trattate con BNT162b2. Le cellule Huh7 sono state trattate
senza (Ctrl) o con 0,5 (V1), 1 (V2) e 2 µg/mL (V3) di BNT162b2 per 6
(punti verdi), 24 (punti rossi) e 48 ore (punti blu) . RNA è stato
purificato e la qPCR è stata eseguita utilizzando primer mirati a LINE-1 . I livelli di RNA di LINE-1 sono presentati come valori di 2 −ΔΔCT relativi ai geni di mantenimento GAPDH e ACTB . I risultati provengono da cinque esperimenti indipendenti ( n = 5). di Student a due code t . I dati sono espressi come media ± SEM. (* p < 0,05; ** p < 0,01; *** p < 0,001 rispetto al rispettivo controllo in ogni momento, o come indicato; † p < 0,05 vs. 6 h-Ctrl).
Successivamente,
abbiamo studiato l'effetto di BNT162b2 sul livello di proteina LINE-1.
La LINE-1 a lunghezza intera è costituita da una regione non tradotta
(UTR) di 5′, due frame di lettura aperti (ORF), ORF1 e ORF2 e una UTR
3′, di cui ORF1 è una proteina legante l'RNA con attività chaperone. È
stato dimostrato che l'attività di retrotrasposizione di LINE-1
coinvolge la traslocazione di ORF1 nel nucleo [ 35 ].
Le cellule Huh7 trattate con o senza BNT162b2 (0,5, 1,0 e 2,0 µg/mL)
per 6 ore sono state fissate e colorate con anticorpi che si legano a
LINE-1 ORF1p e sonda specifica del DNA Hoechst per la visualizzazione
del nucleo cellulare ( Figura ).
La quantificazione dell'intensità della colorazione
dell'immunofluorescenza ha mostrato che BNT162b2 ha aumentato i livelli
di proteina LINE-1 ORF1p sia nell'intera area cellulare che nel nucleo a
tutte le concentrazioni testate ( Figura 4 b-d).
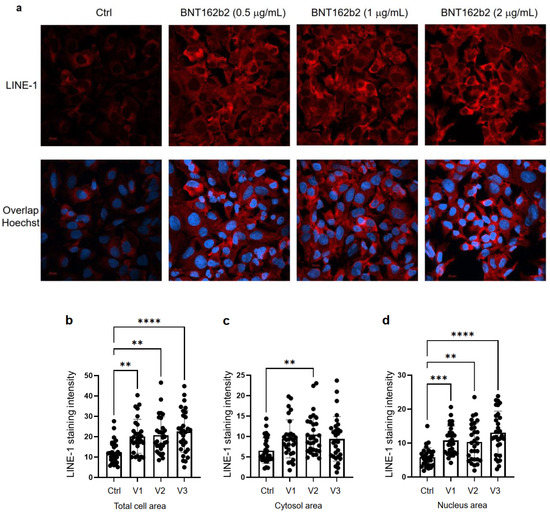
Figura 4.
Immunoistochimica delle cellule Huh7 trattate con BNT162b2 sulla
distribuzione della proteina LINE-1. Le cellule Huh7 sono state trattate
senza (Ctrl) o con 0,5, 1 e 2 µg/mL di BNT162b2 per 6 ore. Le cellule
sono state fissate e colorate con anticorpi che si legano a LINE-1 ORF1p
(rosso) e sonda specifica del DNA Hoechst per la visualizzazione del
nucleo cellulare (blu). ( a ) Immagini rappresentative dell'espressione di LINE-1 in cellule Huh7 trattate con o senza BNT162b2. ( b – d ) Quantificazione della proteina LINE-1 nell'intera area cellulare ( b ), nel citosol ( c ) e nel nucleo ( d ).
Tutti i dati sono stati analizzati utilizzando One-Way ANOVA e i
grafici sono stati creati utilizzando GraphPad Prism V 9.2. Tutti i dati
sono presentati come media ± DS (** p < 0,01; *** p < 0,001; **** p < 0,0001 come indicato).
3.3. Rilevamento del DNA BNT162b2 trascritto inverso nelle cellule Huh7
Uno studio precedente ha dimostrato che l'ingresso della proteina LINE-1 nel nucleo è associato alla retrotrasposizione [ 35 ].
Nell'esperimento di colorazione con immunofluorescenza sopra descritto,
sono stati osservati livelli aumentati di LINE-1 nel nucleo già alla
concentrazione più bassa di BNT162b2 (0,5 µ g/mL). Per esaminare se
BNT162b2 viene trascritto inversamente nel DNA quando LINE-1 è elevato,
abbiamo purificato il DNA genomico da cellule Huh7 trattate con 0,5 µ
g/mL di BNT162b2 per 6, 24 e 48 h. Il DNA purificato è stato trattato
con RNasi per rimuovere l'RNA e sottoposto a PCR utilizzando primer
mirati a BNT162b2, come illustrato nella Figura 1 . I frammenti di DNA amplificati sono stati quindi visualizzati mediante elettroforesi e purificati con gel ( Figura 5 ).
Gli ampliconi del DNA BNT162b2 sono stati rilevati in tutti e tre i
punti temporali (6, 24 e 48 ore). Il sequenziamento di Sanger ha
confermato che gli ampliconi del DNA erano identici alla sequenza
BNT162b2 affiancata dai primer ( Tabella 2 ).
Per garantire che gli ampliconi del DNA fossero derivati dal DNA ma
non dall'RNA BNT162b2, abbiamo anche eseguito la PCR su RNA purificato
da cellule Huh7 trattate con 0,5 µ g/mL di BNT162b2 per 6 ore, con o
senza trattamento con RNasi (Ctrl 5 e 6 in Figura 5 ), e nessun amplicone è stato rilevato nei campioni di RNA sottoposti a PCR.
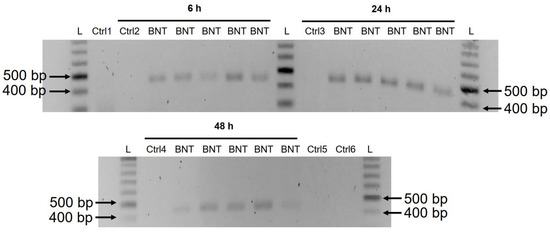
Figura 5.
Rilevazione di ampliconi di DNA di BNT162b2 in cellule Huh7 trattate
con BNT162b2. Le cellule Huh7 sono state trattate senza (Ctrl) o con 0,5
µg/mL di BNT162b2 per 6, 24 e 48 ore. Il DNA genomico è stato
purificato e digerito con 100 µg/mL di RNasi. La PCR è stata eseguita su
tutti i campioni con primer rivolti a BNT162b2, come mostrato nella Figura 1 e Tabella 1 .
Gli ampliconi del DNA (444 bps) sono stati visualizzati su gel di
agarosio. BNT: BNT162b2; L: scala del DNA; Ctrl1: cellule Huh7 in
coltura; Ctrl2: cellule Huh7 senza trattamento BNT162b2 raccolte a 6 h;
Ctrl3: cellule Huh7 senza trattamento BNT162b2 raccolte a 24 ore; Ctrl4:
cellule Huh7 senza trattamento BNT162b2 raccolte a 48 h; Ctrl5: RNA da
cellule Huh7 trattate con 0,5 µ g/mL di BNT162b2 per 6 h; Ctrl6: RNA da
cellule Huh7 trattate con 0,5 µ g/mL di BNT162b2 per 6 ore, digerite con
RNasi.
Tavolo 2.
Risultato del sequenziamento di Sanger dell'amplicone BNT162b2.

4. Discussione
In
questo studio presentiamo prove che il vaccino BNT162b2 dell'mRNA
COVID-19 è in grado di entrare nella linea cellulare del fegato umano
Huh7 in vitro. L'mRNA di BNT162b2 viene trascritto inversamente a
livello intracellulare nel DNA fino a 6 ore dopo l'esposizione a
BNT162b2. Un possibile meccanismo per la trascrizione inversa è
attraverso la trascrittasi inversa endogena LINE-1 e la distribuzione
della proteina del nucleo di LINE-1 è elevata da BNT162b2.
L'accumulo intracellulare di LNP negli epatociti è stato dimostrato in vivo [ 36 ].
Uno studio preclinico su BNT162b2 ha mostrato che BNT162b2 entra nelle
cellule HEK293T della linea cellulare umana e porta a un'espressione
robusta dell'antigene BNT162b2 [ 37 ].
Pertanto, in questo studio, abbiamo prima studiato l'ingresso di
BNT162b2 nelle cellule Huh7 della linea cellulare del fegato umano. La
scelta delle concentrazioni di BNT162b2 utilizzate in questo studio
merita una spiegazione. BNT162b2 viene somministrato come una serie di
due dosi a tre settimane di distanza e ciascuna dose contiene 30 µg di
BNT162b2 in un volume di 0,3 mL, il che rende la concentrazione locale
nel sito di iniezione al massimo di 100 µg/mL [ 31 ].
Uno studio precedente sui vaccini mRNA contro i virus dell'influenza
H10N8 e H7N9 utilizzando un sistema di rilascio LNP simile ha mostrato
che il vaccino mRNA può distribuirsi in modo piuttosto aspecifico a
diversi organi come fegato, milza, cuore, rene, polmone e cervello e la
concentrazione nel il fegato è circa 100 volte inferiore a quello del
sito di iniezione intramuscolare [ 38 ].
Nel rapporto di valutazione su BNT162b2 fornito all'EMA da Pfizer, gli
studi di distribuzione farmacocinetica nei ratti hanno dimostrato che
una proporzione relativamente ampia (fino al 18%) della dose totale si
distribuisce al fegato [ 26 ].
Abbiamo quindi scelto di utilizzare 0,5, 1 e 2 μg/mL di vaccino nei
nostri esperimenti sulle cellule del fegato. Tuttavia, l'effetto di una
gamma più ampia di concentrazioni sempre più basse di BNT162b2 dovrebbe
essere verificato anche in studi futuri.
Nel
presente studio, abbiamo impiegato una linea cellulare di fegato umano
per l'indagine in vitro. Vale la pena indagare se le cellule del fegato
presentano anche la proteina spike SARS-CoV-2 derivata dal vaccino, che
potrebbe potenzialmente rendere le cellule del fegato bersagli per
cellule T citotossiche reattive della proteina spike precedentemente
innescate. Sono stati segnalati casi su individui che hanno sviluppato
epatite autoimmune [ 39 ]
dopo la vaccinazione con BNT162b2. Per ottenere una migliore
comprensione dei potenziali effetti di BNT162b2 sulla funzione epatica,
sono desiderati modelli in vivo per studi futuri.
Nel rapporto sulla tossicità di BNT162b2, non sono stati forniti studi di genotossicità o cancerogenicità [ 26 ].
Il nostro studio mostra che BNT162b2 può essere trascritto inversamente
al DNA nella linea cellulare del fegato Huh7, e questo può dare adito
alla preoccupazione se il DNA derivato da BNT162b2 possa essere
integrato nel genoma dell'ospite e influenzare l'integrità del DNA
genomico, che può potenzialmente mediare genotossicità effetti
collaterali. In questa fase, non sappiamo se il DNA trascritto inverso
da BNT162b2 sia integrato nel genoma cellulare. Sono necessari ulteriori
studi per dimostrare l'effetto di BNT162b2 sull'integrità genomica,
compreso il sequenziamento dell'intero genoma delle cellule esposte a
BNT162b2, nonché i tessuti di soggetti umani che hanno ricevuto la
vaccinazione BNT162b2.
Il
retrotrasposone autonomo umano LINE-1 è una trascrittasi inversa
endogena cellulare e l'unico trasposone attivo rimasto nell'uomo, in
grado di retrotrasporre se stesso e altri elementi non autonomi [ 40 , 41 ] e circa il 17% del genoma umano è composto da sequenze LINE-1 [ 42 ]. Gli Alu ,
gli elementi nucleotidici brevi e intervallati (SINE), il numero
variabile di ripetizioni in tandem (VNTR), nonché gli pseudogeni
cellulari elaborati con mRNA, sono retrotrasposti dalle proteine di
retrotrasposizione LINE-1 che lavorano in trans [ 43 , 44 ].
Uno studio recente ha dimostrato che la LINE-1 endogena media la
trascrizione inversa e l'integrazione delle sequenze SARS-CoV-2 nei
genomi delle cellule umane infette [ 25 ].
Inoltre, l'espressione della LINE-1 endogena è spesso aumentata in caso
di infezione virale, inclusa l'infezione da SARS-CoV-2 [ 45 , 46 , 47 ]. Precedenti studi hanno dimostrato che l'attività di retrotrasposizione della LINE-1 è regolata dal metabolismo dell'RNA [ 48 , 49 ], dalla risposta al danno del DNA [ 50 ] e dall'autofagia [ 51 ].
La retrotrasposizione efficiente della LINEA-1 è spesso associata al
ciclo cellulare e alla rottura dell'involucro nucleare durante la
mitosi. 52 , 53 ], nonché retrovirus esogeni [ 54 , 55 ],
che promuovono l'ingresso della LINE-1 nel nucleo. Nel nostro studio,
abbiamo osservato una maggiore distribuzione di LINE-1 ORF1p determinata
dall'immunoistochimica nel nucleo da BNT162b2 a tutte le concentrazioni
testate (0,5, 1 e 2 μg/mL), mentre un'elevata di LINE-1 espressione
genica concentrazione (2 μg/mL). Vale la pena notare che la
trascrizione genica è regolata dalle modificazioni della cromatina,
dalla regolazione del fattore di trascrizione e dalla velocità di
degradazione dell'RNA, mentre la regolazione traslazionale della
proteina implica il reclutamento di ribosomi sul codone di inizio, la
modulazione dell'allungamento del peptide, la fine della sintesi
proteica o la biogenesi del ribosoma . Questi due processi sono
controllati da meccanismi diversi e pertanto potrebbero non mostrare
sempre gli stessi modelli di cambiamento in risposta a sfide esterne.
L'esatta regolazione dell'attività LINE-1 in risposta a BNT162b2 merita
ulteriori studi.
Il
modello cellulare che abbiamo utilizzato in questo studio è una linea
cellulare di carcinoma, con replicazione attiva del DNA che differisce
dalle cellule somatiche non in divisione. È stato anche dimostrato che
le cellule Huh7 mostrano un'espressione genica e proteica significativa e
diversa, comprese le proteine sovraregolate coinvolte nel metabolismo
dell'RNA [ 56 ].
Tuttavia, la proliferazione cellulare è attiva anche in diversi tessuti
umani come il midollo osseo o gli strati basali degli epiteli, nonché
durante l'embriogenesi, ed è quindi necessario esaminare l'effetto di
BNT162b2 sull'integrità genomica in tali condizioni. Inoltre,
un'efficace retrotrasposizione di LINE-1 è stata riportata anche in
cellule non in divisione e differenziate in modo terminale, come i
neuroni umani [ 57 , 58 ].
Il
rapporto di valutazione Pfizer EMA ha anche mostrato che BNT162b2 si
distribuisce nella milza (<1,1%), nelle ghiandole surrenali
(<0,1%), così come una radioattività bassa e misurabile nelle ovaie e
nei testicoli (<0,1%) [ 26 ].
Inoltre, dal rapporto di valutazione Pfizer EMA non sono disponibili
dati sul trasferimento placentare di BNT162b2. I nostri risultati hanno
mostrato che l'mRNA di BNT162b2 entra prontamente nelle cellule Huh7 a
una concentrazione (0,5 µg/mL) corrispondente allo 0,5% della
concentrazione del sito di iniezione locale, induce cambiamenti nel gene
LINE-1 e nell'espressione proteica e, entro 6 ore, trascrizione inversa
di BNT162b2 può essere rilevato. È quindi importante studiare
ulteriormente l'effetto di BNT162b2 su altri tipi di cellule e tessuti
sia in vitro che in vivo.
5. Conclusioni
Il
nostro studio è il primo studio in vitro sull'effetto del vaccino mRNA
COVID-19 BNT162b2 sulla linea cellulare del fegato umano. Presentiamo
prove sull'ingresso rapido di BNT162b2 nelle cellule e sulla successiva
trascrizione inversa intracellulare dell'mRNA di BNT162b2 nel DNA.
Materiali supplementari
È possibile scaricare le seguenti informazioni di supporto all'indirizzo: https://www.mdpi.com/article/10.3390/cimb44030073/s1 .
Contributi dell'autore
MA,
FOF, DY, MB e CL hanno eseguito esperimenti in vitro. MA e FOF hanno
eseguito l'analisi dei dati. MR e YDM hanno contribuito all'attuazione
della ricerca, progettato e supervisionato lo studio. YDM ha scritto il
documento con il contributo di tutti gli autori. Tutti gli autori
hanno letto e accettato la versione pubblicata del manoscritto.
Finanziamento
Questo
studio è stato sostenuto dallo Swedish Research Council, Strategic
Research Area Exodiab, Dnr 2009-1039, dal Fondo del governo svedese per
la ricerca clinica (ALF) e dalla fondazione dello Skåne University
Hospital.
Dichiarazione del comitato di revisione istituzionale
Non applicabile.
Dichiarazione di consenso informato
Non applicabile.
Dichiarazione sulla disponibilità dei dati
Tutti i dati a supporto dei risultati di questo studio sono disponibili nell'articolo e nelle informazioni di supporto .
Ringraziamenti
Gli
autori ringraziano Sven Haidl, Maria Josephson, Enming Zhang, Jia-Yi
Li, Caroline Haikal e Pradeep Bompada per il loro supporto a questo
studio.
Conflitto di interessi
Gli autori dichiarano assenza di conflitto di interesse.
Riferimenti
- Organizzazione mondiale della Sanità. Cruscotto Coronavirus (COVID-19). Disponibile online: https://covid19.who.int/ (accesso il 22 febbraio 2022).
- Mulligan, MJ; Lyke, KE; Kitchin, N.; Absalon, J.; Gurtman, A.; Lockhart, S.; Neuzil, K.; Raabe, V.; Bailey, R.; Swanson, KA; et al. Studio di fase I/II del vaccino a RNA COVID-19 BNT162b1 negli adulti. Natura 2020 , 586 , 589–593. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Walsh, EE; Frenck, RW, Jr.; Falsey, AR; Kitchin, N.; Absalon, J.; Gurtman, A.; Lockhart, S.; Neuzil, K.; Mulligan, MJ; Bailey, R.; et al. Sicurezza e immunogenicità di due candidati al vaccino COVID-19 a base di RNA. N. ingl. J. Med. 2020 , 383 , 2439–2450. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Polack, FP; Tommaso, SJ; Kitchin, N.; Absalon, J.; Gurtman, A.; Lockhart, S.; Perez, JL; Perez Marc, G.; Moreira, ED; Zerbini, C.; et al. Sicurezza ed efficacia del vaccino BNT162b2 mRNA COVID-19. N. ingl. J. Med. 2020 , 383 , 2603–2615. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Harris, RJ; Hall, JA; Zaidi, A.; Andrews, New Jersey; Dunbar, JK; Dabrera, G. Effetto della vaccinazione sulla trasmissione domestica di SARS-CoV-2 in Inghilterra. N. ingl. J. Med. 2021 , 385 , 759–760. [ Google Scholar ] [ CrossRef ]
- Culo, AA; Omer, Sai Baba; Yan, P.; Shaikh, OS; Mayr, Efficacia del vaccino FB SARS-CoV-2 in una popolazione nazionale ad alto rischio in un ambiente reale. Anna. Stagista. Med. 2021 , 174 , 1404–1408. [ Google Scholar ] [ CrossRef ]
- Dagan, N.; Barda, N.; Mantenuto, E.; Miron, O.; Perchik, S.; Katz, MA; Hernan, MA; Lipsitch, M.; Reis, B.; Vaccino Balicer, RD BNT162b2 mRNA Covid-19 in un ambiente di vaccinazione di massa nazionale. N. ingl. J. Med. 2021 , 384 , 1412–1423. [ Google Scholar ] [ CrossRef ]
- Rossman, H.; Shilo, S.; Meir, T.; Gorfine, M.; Shalit, Stati Uniti; Segal, E. Dinamica COVID-19 dopo un programma nazionale di immunizzazione in Israele. Nat. Med. 2021 , 27 , 1055–1061. [ Google Scholar ] [ CrossRef ]
- Ventilatore, ESSERE; Shen, JY; Lim, XR; Tu, TM; Chang, RCC; Khin, HSW; Koh, JS; Rao, JP; Lau, SL; Abbronzatura, GB; et al. Trombosi venosa cerebrale dopo la vaccinazione con mRNA SARS-CoV-2 BNT162b2: un evento da cigno nero. Sono. J. Hematol. 2021 , 96 , E357–E361. [ Google Scholar ] [ CrossRef ]
- Larson, KF; Ammirati, E.; Adler, ED; Cooper, LT, Jr.; Hong, KN; Saponara, G.; Couri, D.; Cereda, A.; Procopio, A.; Cavalotti, C.; et al. Miocardite dopo vaccinazione BNT162b2 e mRNA-1273. Diffusione 2021 , 144 , 506–508. [ Google Scholar ] [ CrossRef ]
- Menni, C.; Klaser, K.; maggio, A.; Polidori, L.; Capdevila, J.; Louca, P.; Sudre, CH; Nguyen, LH; Disegnato, DA; Merino, J.; et al. Effetti collaterali del vaccino e infezione da SARS-CoV-2 dopo la vaccinazione negli utenti dell'app COVID Symptom Study nel Regno Unito: uno studio osservazionale prospettico. Lancetta infetta. Dis. 2021 , 21 , 939–949. [ Google Scholar ] [ CrossRef ]
- Hansen, T.; Titze, U.; Kulamadayil-Heidenreich, NSA; Glombizza, S.; Tebbe, JJ; Rocken, C.; Schulz, B.; Weise, M.; Wilkens, L. Primo caso di studio post mortem in un paziente vaccinato contro SARS-CoV-2. int. J. Infetta. Dis. 2021 , 107 , 172–175. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Kadali, RAK; Janagama, R.; Perù, S.; Malayala, SV Effetti collaterali del vaccino BNT162b2 mRNA COVID-19: uno studio randomizzato e trasversale con sintomi auto-riferiti dettagliati dagli operatori sanitari. int. J. Infetta. Dis. 2021 , 106 , 376–381. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Parkash, O.; Sharko, A.; Farooqi, A.; Ying, GW; Sura, P. Pancreatite acuta: un possibile effetto collaterale del vaccino COVID-19. Curato 2021 , 13 , e14741. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Mazzatenta, C.; Piccolo, V.; Pace, G.; Romano, I.; Argentiano, G.; Bassi, A. Lesioni purpuriche sulle palpebre sviluppate dopo il vaccino BNT162b2 mRNA COVID-19: un altro pezzo del puzzle cutaneo SARS-CoV-2? J.Eur. Accad. Dermatolo. Venereolo. 2021 , 35 , e543–e545. [ Google Scholar ] [ CrossRef ]
- Lee, EJ; Cine, DB; Gernsheimer, T.; Kessler, C.; Michel, M.; Tarantino, MD; Semplice, JW; Arnold, DM; Godeau, B.; Lambert, parlamentare; et al. Trombocitopenia dopo vaccinazione Pfizer e Moderna SARS-CoV-2. Sono. J. Hematol. 2021 , 96 , 534–537. [ Google Scholar ] [ CrossRef ]
- Isaia, Y.; Kenig, A.; Tsemach-Toren, T.; Amer, R.; Rubin, L.; Hershkovitz, Y.; Kharouf, F. Fenomeni autoimmuni a seguito della vaccinazione SARS-CoV-2. int. Immunofarmaco. 2021 , 99 , 107970. [ Google Scholar ] [ CrossRef ]
- Das, BB; Kohli, Stati Uniti; Ramachandran, P.; Nguyen, HH; Greil, G.; Hussain, T.; Tandon, A.; Kane, C.; Avula, S.; Duru, C.; et al. Miopericardite dopo vaccinazione con mRNA COVID-19 in adolescenti di età compresa tra 12 e 18 anni. J. Pediatr. 2021 , 238 , 26–32.e1. [ Google Scholar ] [ CrossRef ]
- McLaurin-Jiang, S.; Garner, CD; Krutsch, K.; Hale, TW Sintomi materni e infantili dopo la vaccinazione contro il COVID-19 tra le madri che allattano al seno. Allattare. Med. 2021 , 16 , 702–709. [ Google Scholar ] [ CrossRef ]
- Barda, N.; Dagan, N.; Ben Shlomo, Y.; Mantenuto, E.; Waxman, J.; Ohana, R.; Hernan, MA; Lipsitch, M.; Kohane, I.; Netzer, D.; et al. Sicurezza del vaccino BNT162b2 mRNA Covid-19 in un ambiente nazionale. N. ingl. J. Med. 2021 , 385 , 1078–1090. [ Google Scholar ] [ CrossRef ]
- Baden, LR; El Sahly, HM; Essink, B.; Kotloff, K.; Frey, S.; Novak, R.; Diemert, D.; Spector, SA; Rouphael, N.; Creech, CB; et al. Efficacia e sicurezza del vaccino mRNA-1273 SARS-CoV-2. N. ingl. J. Med. 2021 , 384 , 403–416. [ Google Scholar ] [ CrossRef ]
- Sadoff, J.; Grigio, G.; Vandebosch, A.; Cardenas, V.; Shukarev, G.; Grinsztejn, B.; Goepfert, Pennsylvania; Truyers, C.; Fennema, H.; Spiessens, B.; et al. Sicurezza ed efficacia del vaccino monodose Ad26.COV2.S contro Covid-19. N. ingl. J. Med. 2021 , 384 , 2187–2201. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Eichinger, S.; Warkentin, TE; Greinacher, A. Trombocitopenia trombotica dopo la vaccinazione ChAdOx1 nCoV-19. Rispondere. N. ingl. J. Med. 2021 , 385 , e11. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Doroftei, B.; Ciobica, A.; Ilie, OD; Maftei, R.; Ilea, C. Mini-Review Discutendo l'affidabilità e l'efficienza dei vaccini COVID-19. Diagnostica 2021 , 11 , 579. [ Google Scholar ] [ CrossRef ]
- Zhang, L.; Richards, A.; Barrasa, MI; Hughes, SH; Giovane, RA; Jaenisch, R. L'RNA SARS-CoV-2 a trascrizione inversa può integrarsi nel genoma delle cellule umane in coltura e può essere espresso nei tessuti derivati dal paziente. Proc. Natl. Accad. Sci. USA 2021 , 118 , e2105968118. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Disponibile online: https://www.ema.europa.eu/en/documents/assessment-report/comirnaty-epar-public-assessment-report_en.pdf (visitato il 24 febbraio 2022).
- Tanaka, H.; Takata, nord; Sakurai, Y.; Yoshida, T.; Inoue, T.; Tamagawa, S.; Nakai, Y.; Tange, K.; Yoshioka, H.; Maeki, M.; et al. Consegna di oligonucleotidi utilizzando un materiale simile a un lipido autodegradabile. Farmaceutica 2021 , 13 , 544. [ Google Scholar ] [ CrossRef ]
- Sedic, M.; Senn, JJ; Lynn, A.; Laska, M.; Smith, M.; Platz, SJ; Bolen, J.; Hoge, S.; Bulychev, A.; Jacquinet, E.; et al. Valutazione della sicurezza dell'mRNA modificato formulato con nanoparticelle lipidiche nel ratto Sprague-Dawley e nella scimmia Cynomolgus. Veterinario. Patolo. 2018 , 55 , 341–354. [ Google Scholar ] [ CrossRef ]
- Sato, Y.; Matsui, H.; Yamamoto, N.; Sato, R.; Munakata, T.; Kohara, M.; Harashima, H. La consegna altamente specifica di siRNA agli epatociti aggira la tossicità associata alle nanoparticelle lipidiche mediata dalle cellule endoteliali portando alla diminuzione sicura ed efficace del virus dell'epatite B. J. Controllo. Rilascio 2017 , 266 , 216–225. [ Google Scholar ] [ CrossRef ]
- Heidel, JD; Yu, Z.; Liu, JY; Rele, SM; Liang, Y.; Zeidan, RK; Kornbrust, DJ; Davis, ME Amministrazione in primati non umani di dosi endovenose crescenti di nanoparticelle mirate contenenti siRNA della subunità M2 della ribonucleotide reduttasi. Proc. Natl. Accad. Sci. USA 2007 , 104 , 5715–5721. [ Google Scholar ] [ CrossRef ]
- Disponibile online: https://www.cvdvaccine-us.com/ (accesso il 24 febbraio 2022).
- Disponibile online: http://bridgeslab.sph.umich.edu/protocols/index.php/Preparation_of_Tail_Samples_(for_Genotyping) (accesso il 24 febbraio 2022).
- Gallud, A.; Munson, MJ; Liu, K.; Idstrom, A.; Barriga, HM; Tabaei, S.; Aliakbarinodehi, N.; Ojansivu, M.; Lubart, Q.; Douch, JJ; et al. L'evoluzione temporale dell'eliminazione del PEG e dell'incoronazione delle proteine sieriche determina la cinetica di assorbimento cellulare e la consegna di nanoparticelle lipidiche. bioRxiv 2021 . [ Google Scholar ] [ CrossRef ]
- RNA del messaggero dell'Organizzazione mondiale della sanità che codifica per la glicoproteina a punta SARS-CoV-2 a lunghezza intera. 2020. Disponibile online: https://web.archive.org/web/20210105162941/https://mednet-communities.net/inn/db/media/docs/11889.doc (accesso il 24 febbraio 2022).
- Mita, P.; Wudzinska, A.; Sole, X.; Andrade, J.; Nayak, S.; Kahler, DJ; Badri, S.; La Cava, J.; Ueberheide, B.; Yun, CY; et al. Localizzazione della proteina LINE-1 e dinamica funzionale durante il ciclo cellulare. Elife 2018 , 7 , e30058. [ Google Scholar ] [ CrossRef ]
- Sato, Y.; Kinami, Y.; Hashiba, K.; Harashima, H. Diverse cinetiche per l'assorbimento epatico di nanoparticelle lipidiche tra il recettore dell'apolipoproteina E/lipoproteina a bassa densità e la via del recettore N-acetil-d-galattosamina/asialoglicoproteina. J. Controllo. Rilascio 2020 , 322 , 217–226. [ Google Scholar ] [ CrossRef ]
- Vogel, AB; Kanevsky, I.; Che, Y.; Swanson, KA; Muik, A.; Vormehr, M.; Kranz, LM; Walzer, KC; Hein, S.; Guler, A.; et al. I vaccini BNT162b proteggono i macachi rhesus da SARS-CoV-2. Natura 2021 , 592 , 283–289. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Bahl, K.; Senn, JJ; Yuzhakov, O.; Bulychev, A.; Brito, LA; Hassett, KJ; Laska, io; Smith, M.; Almarsson, O.; Thompson, J.; et al. Dimostrazione preclinica e clinica dell'immunogenicità mediante vaccini mRNA contro i virus influenzali H10N8 e H7N9. Mol. Là. 2017 , 25 , 1316–1327. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Bril, F.; Al Difalha, S.; Dean, M.; Fettig, DM Epatite autoimmune in via di sviluppo dopo il vaccino contro la malattia di coronavirus 2019 (COVID-19): causalità o incidente? J. Hepatol. 2021 , 75 , 222–224. [ Google Scholar ] [ CrossRef ]
- Kazazian, HH, Jr.; Moran, JV Mobile DNA in Salute e Malattia. N. ingl. J. Med. 2017 , 377 , 361–370. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- bara, JM; Fan, H. La scoperta della trascrittasi inversa. Annu. Rev. Virol. 2016 , 3 , 29–51. [ Google Scholar ] [ CrossRef ]
- Lander, ES; Linton, LM; Birren, B.; Nusbaum, C.; Zody, MC; Baldwin, J.; Devon, K.; Dewar, K.; Doyle, M.; FitzHugh, W.; et al. Sequenziamento iniziale e analisi del genoma umano. Natura 2001 , 409 , 860–921. [ Google Scholar ] [ CrossRef ]
- Ostertag, EM; Goodier, JL; Zhang, Y.; Gli elementi Kazazian, HH, Jr. SVA sono retrotrasposoni non autonomi che causano malattie negli esseri umani. Sono. J. Hum. Genet. 2003 , 73 , 1444–1451. [ Google Scholar ] [ CrossRef ]
- Hancks, DC; Kazazian, HH, Jr. Retrotrasposoni umani attivi: variazione e malattia. Corr. Opin. Genet. Dev. 2012 , 22 , 191–203. [ Google Scholar ] [ CrossRef ]
- Jones, RB; Canzone, H.; Xu, Y.; Guarnigione, KE; Buzdin, AA; Anwar, N.; cacciatore, DV; Mujib, S.; Mihajlovic, V.; Martin, E.; et al. Il DNA dell'elemento retrotrasposabile LINE-1 si accumula nelle cellule infette da HIV-1. J. Virol. 2013 , 87 , 13307–13320. [ Google Scholar ] [ CrossRef ]
- Macchietto, MG; Langlois, RA; Shen, SS Regolazione dell'espressione di elementi trasponibili indotta da virus nelle cellule ospiti umane e di topo. Scienziato della vita Alleanza 2020 , 3 , e201900536. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Yin, Y.; Liu, XZ; Esadecimale.; Zhou, LQ esogeno Coronavirus interagisce con il retrotrasposone endogeno nelle cellule umane. Davanti. Cellula infetta. microbiolo. 2021 , 11 , 609160. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Belancio, vicepresidente; Roy-Engel, AM; Deininger, P. L'impatto di più siti di giunzione negli elementi L1 umani. Gene 2008 , 411 , 38–45. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Dai, L.; Taylor, SM; O'Donnell, KA; Boeke, la proteina C1 legante JD Poly(A) è essenziale per un'efficiente retrotrasposizione di L1 e influenza la formazione di L1 RNP. Mol. Biol cellulare. 2012 , 32 , 4323–4336. [ Google Scholar ] [ CrossRef ]
- Servo, G.; Streva, Virginia; Derbes, RS; Wijetunge, MI; Neeland, M.; bianco, tubercolosi; Belancio, vicepresidente; Roy-Engel, AM; Deininger, PL Il percorso di riparazione dell'escissione del nucleotide limita la retrotrasposizione L1. Genetica 2017 , 205 , 139–153. [ Google Scholar ] [ CrossRef ]
- Guo, H.; Chitiprolu, M.; Gagnon, D.; Meng, L.; Perez-Iratxeta, C.; Lagace, D.; Gibbings, D. L'autofagia supporta la stabilità genomica degradando l'RNA del retrotrasposone. Nat. Comune. 2014 , 5 , 5276. [ Google Scholar ] [ CrossRef ]
- Xie, Y.; Compagni, L.; Ivics, Z.; Izsvak, Z.; Martin, SL; An, W. La divisione cellulare promuove un'efficiente retrotrasposizione in una linea cellulare reporter L1 stabile. Mob. DNA 2013 , 4 , 10. [ Google Scholar ] [ CrossRef ]
- Shi, X.; Seluanov, A.; Gorbunova, V. Le divisioni cellulari sono necessarie per la retrotrasposizione L1. Mol. Biol cellulare. 2007 , 27 , 1264–1270. [ Google Scholar ] [ CrossRef ]
- Goff, SP Fattori host sfruttati dai retrovirus. Nat. Rev. Microbiol 2007 , 5 , 253–263. [ Google Scholar ] [ CrossRef ]
- Suzuki, Y.; Craigie, R. La strada per la cromatina: ingresso nucleare dei retrovirus. Nat. Rev. Microbiol. 2007 , 5 , 187–196. [ Google Scholar ] [ CrossRef ]
- Shi, J.; Wang, X.; Lyu, L.; Jiang, H.; Zhu, HJ Confronto dell'espressione proteica tra fegati umani e linee cellulari epatiche HepG2, Hep3B e Huh7 utilizzando la proteomica SWATH e MRM-HR: concentrarsi sugli enzimi che metabolizzano i farmaci. Metab farmaco. Farmacocinetta. 2018 , 33 , 133–140. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Kubo, S.; Seleme, MC; Soifer, HS; Perez, JL; Moran, JV; Kazazian, HH, Jr.; Kasahara, retrotrasposizione N. L1 in cellule somatiche umane primarie e non in divisione. Proc. Natl. Accad. Sci. USA 2006 , 103 , 8036–8041. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Macia, A.; Widmann, TJ; Era, RS; Ayllon, V.; Sanchez, L.; Benkaddour-Boumzaouad, M.; Munoz-Lopez, M.; Rubio, A.; Amador-Cubero, S.; Blanco-Jimenez, E.; et al. Retrotrasposizione LINE-1 ingegnerizzata in neuroni umani non in divisione. Ris. genoma 2017 , 27 , 335–348. [ Google Scholar ] [ CrossRef ] [ PubMed ]
Nota dell'editore: MDPI
rimane neutrale per quanto riguarda le rivendicazioni giurisdizionali
nelle mappe pubblicate e nelle affiliazioni istituzionali. |
© 2022 degli autori. Licenziatario MDPI, Basilea, Svizzera. Questo articolo è un articolo ad accesso aperto distribuito secondo i termini e le condizioni della licenza Creative Commons Attribution (CC BY) ( https://creativecommons.org/licenses/by/4.0/ ).


Commenti
Posta un commento